Replicación
Podemos diferenciar tres etapas en el proceso de replicación del ADN:A) Iniciación:
Consiste, básicamente, en el desenrollamiento y apertura de la doble hélice. Se inicia en una región del ADN denominada oriC o punto de iniciación. Es una zona donde abundan las secuencias de bases GATC.
Durante la iniciación se producen varios acontecimientos:
1. El punto de iniciación es reconocido por unas proteínas específicas que se unen a él. Las enzimas helicasas rompen los enlaces de hidrógeno entre las bases nitrogenadas y la doble hélice se abre como una cremallera.
2. Cuando la doble hélice se abre se produce desenrollamiento en esa zona, lo que crea en las zonas próximas unas tensiones que podrían provocar un mayor enrollamiento. La acción de otras enzimas, las girasas y las topoisomerasas, evita esas tensiones rompiendo y soldando de nuevo la hélice de ADN en estos puntos.
3. Las proteínas SSB (del inglés Single Strand Binding-DNA, proteínas de unión a la cadena sencilla) se unen a las hebras molde e impiden que se vuelva a enrollar. Dejan libre la parte de la hebra que lleva las bases, de modo que éstas sean accesibles para otras moléculas.

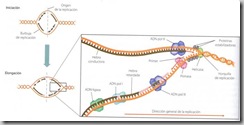
En el lugar de origen de replicación, alrededor de oriC, se ha formado una burbuja de replicación en la que hay dos zonas, con forma de Y, denominadas horquillas de replicación, donde se van a sintetizar las nuevas hebras de ADN. La burbuja de replicación se va extendiendo a lo largo del cromosoma en los dos sentidos, de ahí que se diga que la replicación es bidireccional.


B) Elongación:
Es la fase en la que se sintetiza una nueva hebra de ADN sobre cada hebra de la doble hélice original. Además de las enzimas que actúan en la fase de iniciación, en la elongación intervienen las ADN polimerasas. Hay varios tipos, que se nombran como I, II y III. Su función es doble:
1. Actividad polimerasa: Unen entre sí los nucleótidos que formarán el ADN. Para ello, recorren la hebra molde, seleccionan el desoxirribonucleótido cuya base es complementaria con la de la hebra molde, y lo unen. Las nuevas cadenas de ADN se sintetizan por unión de desoxirribonucleótidos trifosfatos. La energía para el nuevo enlace se obtiene de la hidrólisis de los dos grupos fosfato del nucleótido entrante.
2. Actividad exonucleasa: Eliminan nucleótidos, cuyas bases nitrogenadas están mal apareadas, así como fragmentos de ARN.
Las ADN polimerasas no pueden iniciar de cero la síntesis de una nueva cadena de ADN. Necesitan un fragmento de unos 10 nucleótidos de ARN, denominado cebador o primer, con un extremo hidroxilo 3´ libre al que añadir los nuevos nucleótidos. El cebador es sintetizado por una ARN polimerasa denominada primasa. Una vez comenzada la síntesis, la propia cadena de ADN ya sintetizada actúa como cebador.
Al descubrir el modo de acción de la ADN polimerasa se encontró un obstáculo que impedía explicar cómo se desarrollaba este proceso. Se observó que la ADN polimerasa recorre las hebras molde en sentido 3´- 5´ y va uniendo los nuevos nucleótidos en el extremo 3´ hasta que se forman las hebras replicadas.
Sin embargo las dos cadenas de ADN son antiparalelas, cuando se forma la horquilla de replicación la ADN polimerasa sólo puede sintetizar nucleótidos en uno de los dos sentidos. La síntesis de la nueva hebra orientada en sentido 3´- 5´ se realiza sin interrupciones. A esta hebra se la denomina conductora o líder.
El mecanismo de síntesis de la hebra orientada en sentido 5´- 3´ (hebra retardada) fue descubierto en 1973 por R. Okazaki. Consiste en una síntesis discontinua de pequeños fragmentos de ADN de unos mil nucleótidos (fragmento de Okazaki). Cada uno de los fragmentos requiere de un cebador de ARN sintetizado por la primasa cada ciertos intervalos. La ADN polimerasa va eliminando el cebador y sustituyéndolo por ADN. Finalmente la ADN ligasa suelda todos los fragmentos obtenidos.
| POLIMERASA | EXONUCLEASA | POLIMERIZACIÓN | INICIACIÓN | ||
| Dirección | Función | Dirección | Función | ||
| I | 5´à 3´ 3´à 5´ | Elimina cebador Reparación | 5´à 3´ | Síntesis | No |
| II | 3´à 5´ | Reparación | 5´à 3´ | Síntesis | No |
| III | 3´à 5´ | Reparación | 5´à 3´ | Síntesis | No |
C) Corrección de errores:
Durante la replicación es frecuente que se produzcan errores y se incorporen nucleótidos que no tengan correctamente apareadas sus bases. La ADN polimerasa actúa entonces como exonucleasa y elimina los nucleótidos mal apareados.
Aunque el mecanismo de errores es muy eficiente, a veces queda alguno sin corregir. Esos errores pueden ser importantes en la evolución.
3.7. REPLICACIÓN EN LOS EUCARIOTAS
La replicación del ADN en los organismos eucariontes es muy parecida a la de los procariontes, salvo diferencias derivadas, en parte, de la mayor complejidad del material genético de los eucariotas. Las principales diferencias son:
1. Los cromosomas de eucariotas contienen moléculas de ADN muy largas. Para abreviar el proceso, la replicación se inicia de manera simultánea en varios puntos de cada cromosoma denominados replicones. En Drosophila melanogaster el mayor cromosoma contiene unas seis mil horquillas de replicación, y el proceso dura aproximadamente 3 minutos.
2. Existen 5 tipos de ADN polimerasas (α, β, γ, δ y ε) que se reparten todas las tareas de elongación (replicación de la hebra líder y retardada) y corrección de errores. La γ interviene en la replicación del ADN mitocondrial.
3. En los cromosomas de los organismos eucariotas el ADN se encuentra asociado a las histonas, proteínas básicas que no tienen los procariotas, y que durante la replicación se duplican. Las histonas, junto con el ADN, forman un nucleosoma. Parece ser que los nuevos nucleosomas se incorporan a la hebra retardada, mientras que los viejos se quedan en la conductora.
4. El proceso de replicación del ADN se va completando normalmente hasta llegar al extremo del cromosoma, el telómero. Cuando se elimina el último ARN cebador la hebra retardada quedará incompleta ya que la ADN polimerasa no podrá rellenar el hueco, al ser incapaz de sintetizar en dirección 3´- 5´. Para poder completar esta cadena, la polimerasa necesitaría un extremo hidroxilo 3´ libre donde iniciar un nuevo fragmento. Este hecho hace que el telómero se vaya acortando un poco cada vez que la célula se divide, fenómeno que se asocia a los procesos de envejecimiento y muerte celular.

|
MUERTE CELULAR:
Existen dos formas de muerte celular:
Ø Necrosis o muerte accidental: Se produce cuando la célula sufre un daño grave; por ejemplo, por falta de oxígeno. Los caracteres morfológicos que acompañan este tipo de muerte implican un hinchamiento de la célula y una intensa y rápida alteración de la estructura normal de la membrana plasmática y de los orgánulos citoplasmáticos, incluido el núcleo.
Ø Apoptosis o muerte celular programada: Se trata de una muerte natural, en el curso de la cual las células se autodestruyen en ejecución de un programa genético en el que están implicadas proteínas de efectos antagónicos. Se caracteriza porque se produce una retracción celular, una condensación de la cromatina y su fragmentación en oligonucleosomas (por activación de endonucleasas), y culmina con la formación de protuberancias en la superficie de la célula. La célula se rompe en muchos fragmentos o cuerpos apoptóticos que son fagocitados por los macrófagos.
SÍNTESIS DEL ARN: TRANSCRIPCIÓN
La síntesis del ARN o transcripción ocurre en el interior del núcleo. Como requisitos previos necesita:a) Una cadena de ADN que actúe como molde. De las dos cadenas de nucleótidos que forman el gen, sólo una, la denominada molde, se transcribe realmente, mientras que la otra, llamada informativa, no lo hace.
b) Enzimas. El proceso está catalizado por las ARN-polimerasas. En los procariotas sólo existe una, mientras que en los eucariotas existen tres, las llamadas ARN-polimerasas I (formación del ARNr), II (síntesis de todos los ARNm) y III (ARNt y ARNr de pequeño tamaño).
c) Ribonucleótidos trifosfato de A, G, C y U. Se unen mediante un enlace éster entre el ácido fosfórico situado en la posición 5´ de un ribonucleótido trifosfato y el grupo –OH situado en posición 3´del último ribonucleótido de la cadena de ARN en formación.

EL PROCESO DE LA TRANSCRIPCIÓN:
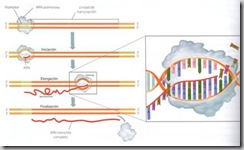
La transcripción consta de 3 etapas: la iniciación, la elongación y la terminación. Tras ella se produce la maduración del ARN.
Ø Iniciación:
Comienza cuando la ARN-polimerasa reconoce en el ADN que se va a transcribir una señal que indica el inicio del proceso. Tales señales, denominadas centros promotores, son unas determinadas secuencias cortas de bases nitrogenadas a las que se une la ARN-polimerasa.
La ARN-polimeras hace que la doble hélice de ADN se abra para permitir que quede expuesta la secuencia de bases del ADN y se puedan incorporar los ribonucleótidos que se van a unir.
Ø Elongación:
Es la adición de sucesivos ribonucleótidos para formar el ARN. La ARN-polimerasa avanza a lo largo de la cadena de ADN “leyéndola” en sentido 3´- 5´, mientras que el sentido de síntesis del ARN es 5´-3´. La enzima selecciona el ribonucleótido trifosfato cuya base es complementaria con la de la cadena de ADN que actúa como molde y lo une, mediante un enlace éster, al siguiente nucleótido, desprendiéndose un grupo pirofosfato (Ppi).
En los eucariotas, tras la unión de los 30 primeros ribonucleótidos se añade en el extremo 5´una “caperuza” formada por metil-guanosin-fosfato, que durante la traducción será una señal de reconocimiento del inicio de lectura.

Ø Terminación:
La ARN-polimerasa reconoce en el ADN unas señales de terminación que indican el final de la transcripción. Esto implica el cierre de la burbuja formada en el ADN y la separación de la ARN-polimerasa del ARN transcrito.
ü En los procariotas, la señal de terminación es una secuencia de bases palindrómicas (secuencias que tienen la misma lectura de izquierda a derecha y de derecha a izquierda) formada por G y C seguidas de varias T, que origina al final del ARN un bucle. Éste favorece su separación del ADN. El bucle se forma por autocomplementariedad de las bases G y C situadas en la cola del ARN.
ü En los eucariotas, la ARN-polimerasa transcribe regiones de ADN largas, que exceden en la longitud de la secuencia que codifica la proteína. En ciertos puntos, una enzima corta el fragmento de ARN que lleva la información para sintetizar la proteína del ARN que sigue transcribiéndose. La señal de corte es una secuencia (AAUAA) que aparece sobre el ARN unos pocos nucleótidos antes del punto de corte, además de otras secuencias mal conocidas. Con posterioridad a la separación del ARN, una enzima (poli-A polimerasa) añade en el extremo final 3´ una secuencia formada por unos 200 nucleótidos de adenina, llamada cola poli-A, que al parecer interviene en los procesos de maduración y transporte del ARN fuera del núcleo.

MADURACIÓN DEL ARN:
A veces, los ARNm no se pueden traducir directamente en proteínas, sino que necesitan un procesamiento previo o maduración postranscripcional.
Organismos procariotas:
 El ARNm de los procariotas puede ser directamente traducido y a partir de él se forma una proteína funcional. No se puede hablar, por tanto, de una maduración de los mensajeros en estos organismos.
El ARNm de los procariotas puede ser directamente traducido y a partir de él se forma una proteína funcional. No se puede hablar, por tanto, de una maduración de los mensajeros en estos organismos. Sin embargo, cuando se transcribe el ADN que codifica los ARNt y los ARNr se forma una larga molécula de ARN que contiene numerosas copias de las secuencias del ARNr o el ARNt. Esta larga molécula, el transcrito primario, es posteriormente cortada en fragmentos más pequeños por enzimas específicas, para dar lugar a los distintos ARNt y ARNr.
Organismos eucariotas:
En los eucariotas la maduración es más compleja, ya que la mayor parte de los genes que codifican las proteínas están fragmentados. Cada gen consta de varios fragmentos denominados intrones y exones, intercalados unos con otros.
ü Los intrones son secuencias de bases más o menos largas que se transcriben, pero que no se traducen, es decir, no codifican una secuencia de aminoácidos.
ü Los exones son las secuencias que se transcriben y se traducen, es decir, tienen información para formar una cadena polipeptídica.
Así pues, el ARN transcrito primario está formado por intrones y exones. Su maduración consiste en la eliminación de los primeros y la unión de los segundos mediante un mecanismo que se conoce con el nombre de splicing (empalme). Requiere la presencia de una enzima llamada ribonucleoproteína proteica nuclear (RNPpn). El proceso de splicing comienza cuando las secuencias intrónicas forman unos bucles que provocan el acercamiento de los extremos de los exones y continúa con el corte de los intrones y la unión de los exones, para formar un ARNm que ya está en condiciones de salir del núcleo.

Los intrones no existen en procariotas y no se sabe que función cumplen en eucariotas. Lo que sí se sabe es que, a veces, un mismo gen puede madurar de diferentes maneras, dependiendo de cómo se eliminen los intrones. De este modo, a partir de un solo gen se pueden obtener diferentes proteínas.
Actualmente se piensa que los genes del primitivo antecesor común a procariotas y eucariotas debía tener intrones. Las bacterias los habrían perdido por selección natural, pues para ellas es crucial dividirse muy rápidamente. Se habrían conservado en eucariotas porque presenta ventajas evolutivas. Las levaduras, que son unos eucariotas con un modo de vida similar al de muchas bacterias, no presentan intrones; sin embargo las mitocondrias, que se cree que descienden de bacterias endosimbiontes, sí tienen intrones en su ADN, pues no están sometidas a la misma presión.
EXPRESIÓN DEL MENSAJE GENÉTICO: TRADUCCIÓN

El núcleo contiene la información genética; esto es, la información necesaria para que se puedan realizar las funciones celulares.
La transmisión de la información genética de los ascendientes a los descendientes y de una generación celular a la siguiente se realiza a través del núcleo celular. Debido a esto en el núcleo se realizará el proceso de duplicación o replicación del ADN ya estudiada.
Los procesos de síntesis del ARN (anteriormente vistos), transcripción de la información genética para la posterior síntesis de proteínas en el hialoplasma, se dan también en el núcleo.
Por último, esta información se traducirá (Traducción) en el citoplasma celular, pues en él se realizará la síntesis de proteínas.

Para que tenga lugar el proceso de traducción o síntesis de proteínas se necesitan:
ü Ribosomas, donde se realiza la síntesis proteica.
ü ARN mensajero, que lleva la información para sintetizar cada proteína.
ü Aminoácidos, que son los componentes de las proteínas.
ü ARN de transferencia, que aporta los aminoácidos en el orden preciso.
ü Enzimas y energía, necesarias en toda reacción de biosíntesis.
La traducción se realiza en los ribosomas, orgánulos citoplasmáticos formados por dos subunidades, una pequeña y otra grande, formadas por ARNr específicos y por proteínas. En la subunidad pequeña se une el ARNm, mientras que en la subunidad grande es donde se unen los aminoácidos para formar la cadena polipeptídica. Ambas se unen cuando van a sintetizar proteínas.
En el ribosoma se distinguen tres lugares diferentes de unión a los ARN de transferencia; el sitio P (peptidil), donde se sitúa la cadena polipeptídica en formación; el sitio A (aminoacil), donde entran los aminoácidos que se van a unir a la cadena proteica, y el sitio E, donde se sitúa el ARNt antes de salir del ribosoma.
ACTIVACIÓN DE LOS AMINOÁCIDOS:
La activación consiste en la unión de un aminoácido con el ARNt que le corresponde. Interviene la enzima aminoacil-ARNt-sintetasa, que da lugar a un complejo denominado aminoacil-ARNt. La reacción requiere energía, que es aportada por la molécula de ATP:
Aminoácido + ATP + Enzima + ARNt à Aminoacil-ARNt + Ppi + AMP + Enzima
La unión del aminoácido y su ARNt se realiza entre el grupo carboxilo del aminoácido y el grupo –OH del extremo 3´ del ARNt.
Existen al menos 20 aminoacil-ARNt-sintetasas, una para cada aminoácido. Estas enzimas son muy específicas, pues han de unir cada aminoácido a los ARNt que les corresponde. Así pues, estas enzimas son piezas clave en la cadena de transferencia de la información.

SÍNTESIS DE PROTEÍNAS:
Excepto con pequeñas diferencias, la síntesis proteica transcurre de igual forma en procariotas y eucariotas. El proceso se puede dividir en varias etapas:
1. Iniciación de la cadena proteica:
La síntesis se inicia cuando la subunidad pequeña del ribosoma y el ARNm se unen en un punto localizado cerca del codón AUG, que es el codón iniciador y marca el inicio de la proteína. A continuación entra en el sitio P del ribosoma un primer aminoacil-ARNt, aquel cuyo anticodón esté formado por 3 bases (UAC) complementarias del codón iniciador. Este primer ARNt lleva unido el aminoácido N-formil metionina (f-Met) en las bacterias, mientras que en los eucariotas es la Metionina (Met). Todas las proteínas inician su síntesis con uno de estos aminoácidos, aunque en algún caso puede separarse una vez formada la proteína.
La subunidad pequeña del ribosoma, el ARNm y el primer aminoacil-ARNt forman el complejo de iniciación, al que con posterioridad se une la subunidad grande del ribosoma.


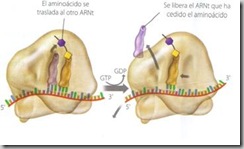
2. Elongación:
La elongación consiste en el alargamiento de la cadena proteica y se inicia cuando un segundo aminoacil-ARNt, cuyo anticodón es complementario al codón situado a continuación del iniciador, “entra” en el ribosoma y ocupa el sitio A que se halla libre.
El siguiente paso es la formación de un enlace peptídico entre el aminoácido que ocupa el sitio P (que suele ser la metionina o formil-metionina) y el nuevo aminoácido que ocupa el sitio A. La reacción de formación del enlace peptídico está catalizada por la enzima peptidil-transferasa, cuya actividad catalítica reside en el ARN que forma parte de esta subunidad, lo que ha llevado a pensar que esta enzima es una ribozima.
Como consecuencia de la formación de este enlace peptídico, el segundo ARNt queda unido por un extremo al dipéptido formado y por el otro a su codón complementario. A continuación se produce la translocación del ribosoma. La translocación implica el desplazamiento del ribosoma a lo largo del ARNm en sentido 5´à 3´. Como este desplazamiento es exactamente de tres bases, el primer ARNt abandona el ribosoma, y el peptidil-ARNt, que todavía se mantiene unido a su codón, pasa a ocupar el sitio P, quedando el sitio A libre.
En estas condiciones otro aminoacil-ARNt se puede incorporar al sitio A, de manera que el proceso de alargamiento de la cadena proteica puede continuar, repitiéndose el ciclo.

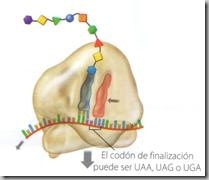
3. Terminación:
 La terminación de la cadena proteica tiene lugar cuando el ribosoma llega a un lugar del ARNm donde se encuentra un codón de terminación (UAA, UAG o UGA), que no es reconocido por ningún ARNt y sí por unos factores de liberación de naturaleza proteica que se sitúan en el sitio A y hacen que la peptidil-transferasa separe, por hidrólisis, la cadena polipeptídica del ARNt.
La terminación de la cadena proteica tiene lugar cuando el ribosoma llega a un lugar del ARNm donde se encuentra un codón de terminación (UAA, UAG o UGA), que no es reconocido por ningún ARNt y sí por unos factores de liberación de naturaleza proteica que se sitúan en el sitio A y hacen que la peptidil-transferasa separe, por hidrólisis, la cadena polipeptídica del ARNt. Una vez completada la traducción, la proteína formada, el ARNm y el ARNt abandonan el ribosoma, que se disocia en sus dos subunidades hasta el momento en que se inicie una nueva síntesis.
A medida que se van sintetizando, las proteínas adquieren la estructura secundaria y terciaria que les corresponde, mediante la formación de enlaces de hidrógeno y enlaces disulfuro entre los aminoácidos que la forman.
Tanto en procariotas como en eucariotas, si el ARNm que se tiene que traducir es lo suficientemente largo, puede ser leído por más de un ribosoma a la vez, formando un polirribosoma o polisoma, que es observable con ayuda del microscopio electrónico.

En los procariotas, al no haber división entre núcleo y citoplasma, la traducción es simultánea a la transcripción: el ARNm comienza a traducirse antes de que termine la transcripción. Las tres etapas se caracterizan la síntesis de proteínas requieren energía, que se obtiene a partir de la liberada en la hidrólisis del GTP a GDP.
CÓDIGO GENÉTICO
El CÓDIGO GENÉTICO es la clave que relaciona la secuencia de bases del ADN o del ARN con la secuencia de aminoácidos en las proteínas.Si el lenguaje cifrado reside en el ADN, es lógico pensar que está basado en la secuencia de bases. Como son cuatro bases (Adenina, Guanina, Timina, Citosina), si las ordenamos de 2 en 2 (V'4,2 = 42 = 16) resultarían 16 variantes, insuficientes ya que existen 20 aminoácidos. Si tomamos variaciones de 3 en 3 (V'4,3 = 43 = 64), resultan 64, es decir, nos sobran tripletes para nombrar a los 20 aminoácidos, pero se demostró que muchos aminoácidos responden a más de un triplete y que hay tripletes que no llaman a ningún aminoácido, los cuales se conocen como “stop, paro o sin sentido”.
El descifrado del código se inició en 1965, se debe a Severo Ochoa (Premio Nobel 1959), que utilizó la enzima polinucleótido-fosforilasa, la cual permitió unir nucleótidos y formar polinucleótidos. Unió varios nucleótidos con la base U y repitiendo esta secuencia varias veces, en presencia de distintos aminoácidos, comprobó que siempre se formaba un polipéptido de fenilalanina. Estaba claro que el triplete UUU llamaba al aminoácido fenilalanina.
Experiencias similares permiten descubrir que el triplete AUG llamaba a la metionina, etc. De este modo se descifró todo el código genético, es decir, los tripletes o codones específicos para los distintos aminoácidos. Sin lugar a duda, fue el mayor avance de los años ´60.
CARACTERÍSTICAS DEL CÓDIGO
1) Es universal. Es válido para todos los seres vivos. Gracias a la genética molecular, se ha descubierto que tiene excepciones, concretamente mitocondrias y algunos protozoos, utilizan un código genético ligeramente diferente.
2) Disposición lineal, cada tres nucleótidos corresponden a un aminoácido específico.
3) Existe un codon de iniciación AUG y tres de terminación UAG, UAA y UGA llamados codones sin sentido, de paro o stop. El codon AUG al mismo tiempo sirve para codificar el aminoácido Metionina. Por tanto todas las proteínas comienzan por la Metionina. Ahora bien, posteriormente, esta Metionina que ocupa la posición inicial puede ser eliminada.
4) El código está degenerado, ya que exceptuando el Triptófano y la Metionina, existen dos o más codones para cada aminoácido. Ello es así puesto que el número de tripletes es superior al de aminoácidos existentes en las proteínas. Los distintos codones que codifican para un mismo aminoácido se denominan codones sinónimos; esto supone una ventaja, ya que en el caso de que se produzcan cambios en algún nucleótido, es decir, que haya mutaciones, no se tiene por qué alterar el orden de los aminoácidos que forman una proteína.


REGULACIÓN DE LA ACCIÓN DE LOS GENES: HIPÓTESIS DEL OPERÓN
 Todas las células de un organismo pluricelular, excepto los gametos, poseen la misma información genética. Ahora bien, no todos los genes se encuentran activos durante el ciclo celular. Uno de los modelos de regulación de la expresión génica mejor conocidos en procariotas es el modelo del operón, descrito en los años cincuenta por Jacob y Monod en Escherichia coli. Un operón se compone de los siguientes elementos:
Todas las células de un organismo pluricelular, excepto los gametos, poseen la misma información genética. Ahora bien, no todos los genes se encuentran activos durante el ciclo celular. Uno de los modelos de regulación de la expresión génica mejor conocidos en procariotas es el modelo del operón, descrito en los años cincuenta por Jacob y Monod en Escherichia coli. Un operón se compone de los siguientes elementos: Ø Promotor (p): Es una secuencia de nucleótidos del ADN a la que se une la ARN polimerasa para iniciar la transcripción de un gen o un conjunto de genes.
Ø Genes estructurales: Aquellos que codifican la síntesis de proteínas implicadas en un mismo proceso metabólico. Se transcriben sin interrupción, de modo que el ARNm resultante lleva la información para varias proteínas y recibe el nombre de ARNm policistrónico.
Ø Operador (o): Secuencia de nucleótidos situada entre el promotor y los genes estructurales.
Ø Gen regulador (r): Puede estar situado en cualquier lugar del cromosoma bacteriano y codifica la proteína que actúa de represor. Cuando la proteína represora se asocia al operador impide físicamente que la ARN-polimerasa se pueda unir al ADN y con ello imposibilita la transcripción. Cuando el represor se separa, la transcripción ya es posible.
Muchos genes no actúan nunca y otros actúan sólo en determinados momentos, pudiendo permanecer durante largos periodos de tiempo inactivos. Para poder comprender el mecanismo de acción de los genes veamos a continuación estos dos modelos de regulación:
REGULACIÓN DE LA ACTUACIÓN DEL OPERÓN LAC EN LA BACTERIA ESCHERICHIA COLI
La ß-galactosidasa es una enzima que rompe el enlace O-glicosídico entre la galactosa y la glucosa en la lactosa.
Si no hay lactosa en el medio, E. coli apenas dispone de unas pocas moléculas de enzima, una o dos solamente.
Sin embargo, si añadimos lactosa al medio donde se encuentra la bacteria, al cabo de unos pocos minutos los niveles de ß-galactosidasa suben hasta alcanzar las 5000 moléculas por célula, aproximadamente.
Aparecen además otras dos enzimas: una permeasa que facilita la absorción de la lactosa a través de la membrana plasmática de la célula y una transacetilasa, necesaria también para el metabolismo de la lactosa.
Jacob y Monod interpretaron estos resultados planteando la hipótesis del operón. Según esta hipótesis la actividad de varios genes que codifican enzimas relacionadas entre sí, genes estructurales, sería desencadenada por la acción de un gen operador, contiguo a los genes estructurales en la molécula de ADN. El conjunto formado por los genes estructurales y el gen operador recibe el nombre de operón.
Si el gen operador se encuentra libre, los genes estructurales se transcriben. A su vez, el gen operador estaría controlado por un gen regulador, que puede estar situado lejos del operón.
Este gen va a sintetizar un ARNm que servirá para la síntesis de una proteína: el represor. Si el represor se encuentra activo se unirá al gen operador inhibiéndolo, con lo que los genes estructurales no se transcribirán.
El operón LAC en E. coli constaría de tres genes estructurales que codificarían respectivamente: la ßgalactosidasa (gen z), la permeasa (gen y) y la transacetilasa (gen a).
Si no hay lactosa en el medio, el gen regulador se traduciría en una proteína, el represor, con dos centros activos. Por uno de ellos sería capaz de unirse al gen operador inhibiendo la síntesis de los ARNm codificados por los genes estructurales z, y, a. Por el otro centro activo podría unirse a la lactosa cuando la hubiese.
La lactosa cambiaría la estructura del represor inactivándolo e impidiendo que éste pudiese unirse al gen operador. De esta manera los genes estructurales se transcribirían produciéndose la síntesis de las tres enzimas que metabolizan la lactosa en E. coli.


LOS OPERONES REPRIMIBLES
Un ejemplo de este tipo de operones es la regulación de los genes responsables de los procesos de síntesis.
Supongamos que la célula necesita producir una determinada cantidad de una sustancia A y que no interesa que haya un exceso de A ni que ésta falte. Supongamos también que para sintetizar A se necesitan tres enzimas: a, b y c.
En estos casos, la proteína que actúa como represor del gen operador se encuentra normalmente en estado inactivo, permitiendo que los genes a, b y c se transcriban y que A se sintetice. Cuando A alcanza unos niveles elevados, se une al represor, activándolo.
El represor activo se une al operador y los genes estructurales a, b y c no se transcriben. Esto hace descender la cantidad de A, con lo que el represor vuelve a estar inactivo, los genes estructurales vuelven a traducirse y vuelve a sintetizarse A.
De esta manera la célula mantiene unas determinadas cantidades de A.
Como se ve, se trata de un mecanismo que funciona como un termostato, manteniendo unos niveles adecuados de una determinada sustancia, en este caso A, necesaria para la célula.

Un ejemplo sería el del “operón del triptófano”:
Funcionamiento del operón “trp” (sin triptófano)
El gen regulador codifica continuamente un represor inactivo que no se une al operador y la ARN-polimerasa puede pasar y transcribir los genes estructurales que codifican la vía metabólica del triptófano.
Funcionamiento del operón «trp» (con triptófano)
El triptófano se une al represor inactivo y modifica su estructura con lo que puede unirse al operador y no deja pasar la ARN-polimerasa: la transcripción cesa.

No hay comentarios:
Publicar un comentario